R 내부데이터, 공공데이터 등 여러 데이터들을 분석해보니 여러가지 함수들을 사용하게 되었습니다. 분석에 사용했던 함수들을 모수/비모수 혹은 범주형/연속형 에 따라 분류하여, 데이터에 따라 적당한 방법을 사용할 수 있는 방법을 작성하겠습니다.
Y : 연속형 ~ X : 범주형 (2그룹)
모수 : T - test [ 두 집단의 평균값 차이 ]
data = PlantGrowth
shapiro.test(data$weight)
data$gp = ifelse(data$group == "ctrl",1,0)
var.test(weight~gp, data)
t.test(weight~gp,data,var.equal=T)- var.test 결과 : p-value > 0.05 이므로, 분산이 같다.
- t.test 결과 : p-value > 0.05 이므로, 두 그룹의 평균 차이는 통계적으로 유의미하지 않다.

비모수 : wilcox - test [ 두 집단의 중심 차이 : 순위 기반 비교]
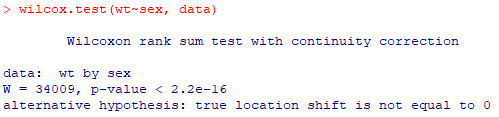
wilcox.test(wt~sex, data)
aggregate(wt~sex,data,summary)- wilcox.test 결과 : p-value < 0.05 이므로, 두 값의 중앙값 차이가 통계적으로 유의미하다.
- mean-range table : 얼마나 차이나는지 보인다.

Y : 연속형 ~ X : 범주형 (3그룹)
모수 : ANOVA test [ 세 집단 이상의 평균값 차이 ]
data = PlantGrowth
by(data$weight, data$group, shapiro.test)
bartlett.test(weight~group, data)
ret = aov(weight~group, data)
summary(ret)
TukeyHSD(ret)- shapiro.test 결과 : 정규성을 만족여부

- bartlett.test 결과 : p-value > 0.05 이므로, 그룹간 분산이 동일하다.

- aov 결과 : p-value < 0.05 이므로, 그룹간 평균차이가 통계적으로 유의미하다.

- TukeyHSD 결과 : diff - 두 그룹의 평균 차이, plot결과 0을 포함하면 그룹간 평균차이가 통계적으로 유의미하지 않음


비모수 : Kruskal [ 세 집단 이상의 중앙값 차이 ]
data = mtcars
data$cyl = as.factor(data$cyl)
by(data$mpg, data$cyl, shapiro.test)
ret = kruskal.test(mpg~cyl, data)
ret = pairwise.wilcox.test(data$mpg, data$cyl, p.adjust.method="bonferroni", exact=FALSE)
ret
boxplot(mpg ~ cyl, data = mtcars,
+ main = "Miles per Gallon by Cylinder",
+ xlab = "Number of Cylinders",
+ ylab = "Miles per Gallon",
+ col = c("lightblue", "pink", "lightgreen"))
> col = c("lightblue", "pink", "lightgreen"))- shapiro.test 결과 : 모두 p-value > 0.05 이므로, 정규분포한다. 그러나, 데이터 수가 적으므로 해당 테스트를 진행한다.

- kruskal.test 결과 : p-value < 0.05 이므로, 그룹 간 중앙값 차이가 통계적으로 유의미하다.

- pairwise.wilcox.test 결과 : 사후검정 결과, 모두 p-value < 0.05 이므로, 중앙값 차이가 유의미하다. plot을 통해 확인한다.


Y : 연속형 ~ X : 연속형
정규분포 가정 : Pearson 상관분석 [ 두 연속형 변수 간의 선형 상관관계 측정 ]
- 두 변수 간의 관계가 선형적이어야함
data = cars
shapiro.test(data$speed)
shapiro.test(data$dist)
cor.test(data$speed, data$dist, method="pearson")
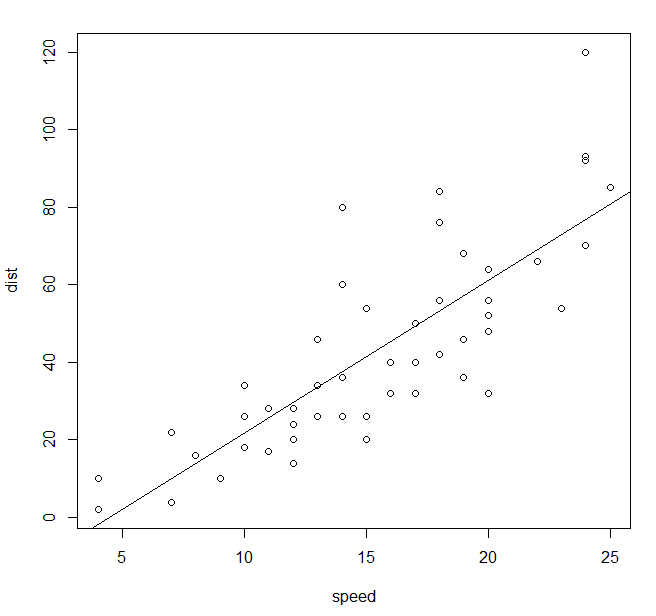
plot(data)
lines(lowess(data))
// 선형회귀
ret = lm(dist~speed, data)
summary(ret)
abline(ret)- shapiro.test 결과 : 모두 p-value > 0.05 이면 pearson 정상적으로 사용, 해당 데이터에선 정규분포라 가정.

- cor.test 결과 : p-value < 0.05 이므로, 상관관계가 통계적으로 유의미. r > 0 이므로 강력한 양의 상관관계 가짐


- plot -> lines : 경향선을 볼 수 있음

- lm 결과 : R-squared = 0.6511 이므로, speed가 dist의 약 65%를 설명한다.
> Residuals : 잔차는 0에 가까울 수록 모델의 예측력이 좋음을 의미함
> Coefficients : Estimate ( 변수가 1 오를 때, y가 얼마나 오르는가 ), Pr ( < 0.05 이면 통계적으로 유의하다 )
> Multiple R-squared : 독립변수가 종속변수의 변동을 얼마나 설명하는가
> F-statistic : p-value < 0.05 이면 모델 전체가 통계적으로 유의미하다.

- plot -> abline : 추세선
가정 요구하지 않음 : Spearman 상관분석 [ 두 변수의 비선형 관계 또는 순위 기반 관계 측정 ]
- 데이터를 순위로 변환한 후 상관계수 계산
data = iris
shapiro.test(data$Sepal.Length)
shapiro.test(data$Petal.Length)
cor.test(data$Sepal.Length, data$Petal.Length, method="spearman", exact=FALSE)
plot(data$Sepal.Length, data$Petal.Length)
lines(lowess(data$Sepal.Length, data$Petal.Length))
// 선형회귀
ret = glm(Petal.Length ~ Sepal.Length, data, family="gaussian")
summary(ret)
plot(data$Sepal.Length, data$Petal.Length)
abline(ret)- shapiro.test 결과 : 정규분포 여부

- cor.test 결과 : spearman test 결과
> p-value < 0.05 이므로, 두 변수간 관계가 통계적으로 유의미함
> rho = 0.881.. 이므로, 두 변수는 강한 양의 단조 관계임

- plot : 비선형 경향성 보임

- glm : 선형회귀
> Coefficients : Estimate ( 꽃바침 길이 1 증가시 꽃잎 길이 1.85 증가 ), Pr ( < 0.05 이므로, 통계적으로 유의미한 영향 )
> AIS : 값이 작을수록 모델이 데이터를 더 잘 설명함

- plot > abline : 추세선

Y : 범주형 ~ X : 범주형
Crosstable + Chisq.test
data = mtcars
CrossTable(data$vs, data$gear)
chisq.test(data$vs, data$gear)
ret = glm(vs~gear, data, family=binomial)
summary(ret)
round(exp(cbind(coef(ret), confint(ret))), 3)- CrossTable 결과 : 변수간 분포를 시각적으로 확인함
> vs = 1 일때, 4단 기어의 비율이 높다 등.. 해석 가능

- chisq.test 결과 : p-value < 0.05 이므로, vs와 gear는 독립적이지 않음 ( 통계적으로 유의미한 관계가 있음 )

- glm 결과 : 로지스틱 회귀
> Coefficient :
Estimate ( 기어 수 1증가할때 로그 오즈의 변화량 )
Std.Error ( 표준오차. 작은수록 신뢰도 놓음 )
z value ( z값 클수록 종속변수에 더 강한 영향 미침 )
Pr ( p-value > 0.05 이므로, 통계적으로 유의하지 않음 )

- exp 결과 : odds 값 얻기
> 기어수 1 증가하면 직렬엔진일 오즈가 1.788배 증가함. ( 기어수 증가할 수록 직렬엔진일 확률이 증가함 )
> 신뢰수준 1을 포함하므로, gear의 효과는 통계적으로 유의미하지 않음

적절한 모델 찾기
data = mtcars
ret = glm(vs ~ wt + hp + factor(gear) + drat, data, family = binomial)
vif(ret)
step(ret)
ret2 = glm(formula = vs ~ hp, family = binomial, data = data)
lrtest(ret,ret2)
summary(ret2)- vif 결과 : 다중공선성 확인 ( 10보다 크면 서로 영향을 많이 주므로, 없애야함 )
- step 결과 : 자동으로 AIC 가장 낮은 모델을 골라줌
- lrtest 결과 : p-value > 0.05 이므로, 두 모델 간 성능 차이가 유의미하지 않다. 즉 새로운 모델을 채택한다.

- summary 결과 : 최종 모델 확인
'R' 카테고리의 다른 글
| R을 활용한 외부데이터 분석 (1) | 2024.10.20 |
|---|---|
| R을 활용한 공공데이터 분석 (6) | 2024.10.20 |

